カルパインは蛋白質のどこを切る?
― カルパインに切断されるタンパク質の切断部位のルール(基質特異性)の解明に向けて ―
カルパインは、多くのタンパク質に対してごく少数の部位を切断します(部位分解といいます)。タンパク質をバラバラに分解するオートファジーで働くプロテアーゼやユビキチン・プロテアソーム系とは、大きく異なります。しかもその切断位置はいつも決まっているので、適当に切っているわけではありません。
カルパインがタンパク質の何を認識して切断位置を決めているかについては、古くから多くの研究がありますが、未だに決定打がない状態です。最も良く解析されているのは、これまでに報告された、カルパインによって切断される基質タンパク質の配列を、切断部位に揃えて全て並べてみて、特定の位置に特定のアミノ酸の来ることが多いとか少ないとかを調べたものです。これを部位特異的配列マトリックス(position-specific sequence matrix, PSSM)と呼ばれます。PSSMをイメージで示したものが下図です(ここでは132種の基質タンパク質からの367箇所の切断部位を並べて解析しました)。
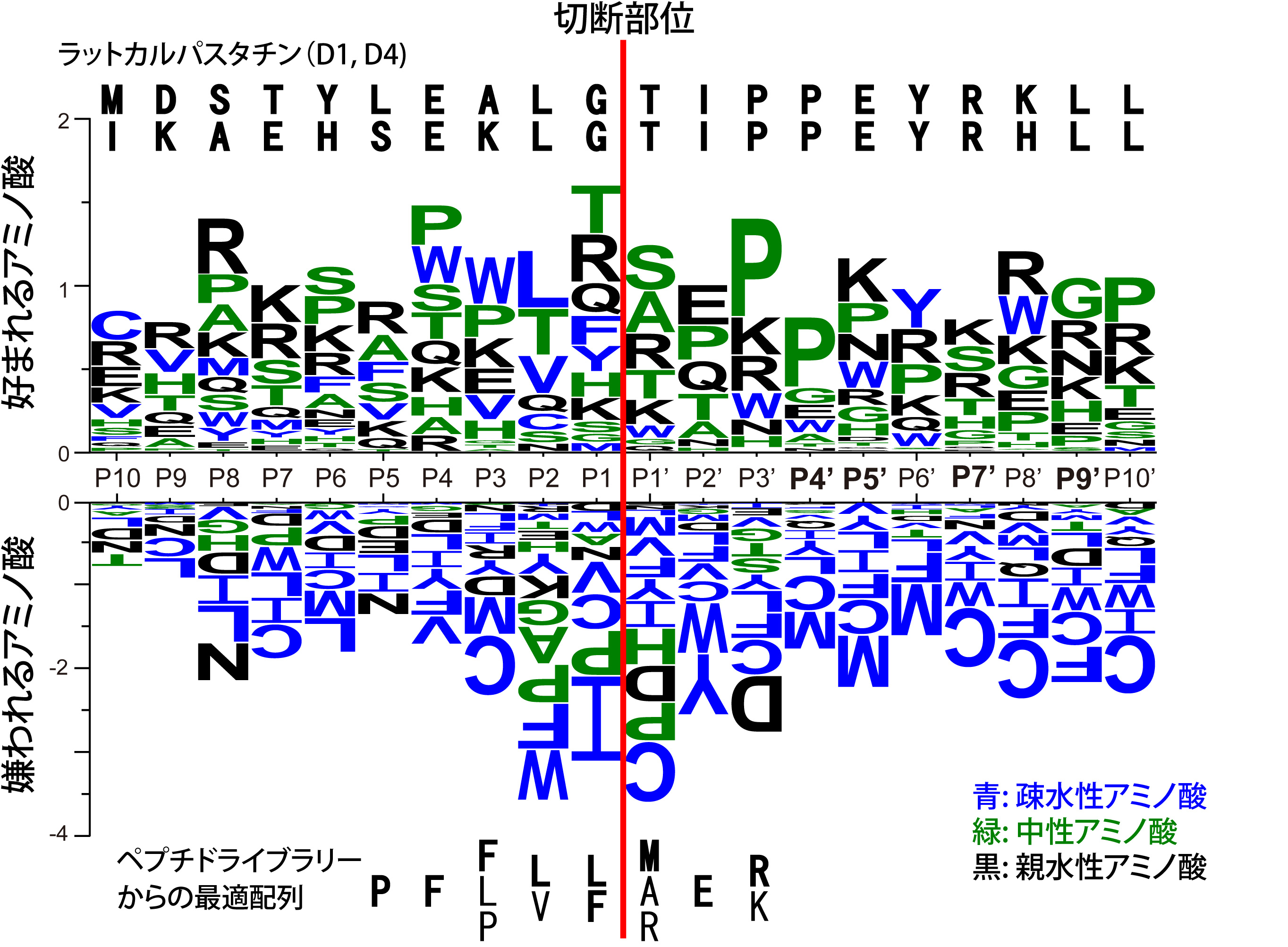
赤線の部位でカルパインは基質タンパク質を切断します。そこからN末端側の方向に向かってアミノ酸残基の位置を逆順にP1、P2、P3…というように呼びます。一方、C末端側の方向に向かっては、P1’、P2’、P3’…というように呼びます。各部位に存在するアミノ酸残基の割合を標準的なタンパク質のアミノ酸組成(タンパク質データベースに存在する全てのタンパク質に現れるアミノ酸残基の割合)で割って、logをとったものが各アミノ酸のアルファベットの長さになっています。つまり、上側(+)に大きく伸びているものほどその部位に偏って多く存在しており、下側(-)に伸びるものほど、その部位には存在しにくいと言うことです。ちょうど平均的に存在するものは大きさが0になります(1の大きさを持つものは標準の2倍(+)または1/2倍(-)の頻度という事になります)。
一番頻度の高いものをとると、CRRKSRPWLT/SEPPKYKRGP(/は切断位置)となりますので、もしこの配列があれば、カルパインは/の位置でとても良くこのタンパク質を切断するように思われます。しかしながら、一番ではなくてもいくつか頻度の高いアミノ酸があるため、なかなか正確に切断部位を予想することは難しいわけです。コンピュータを使って上のデータを利用することにより、カルパインによる切断部位予測を作ることは可能です。各部位(上記ではP10~P10’の20箇所)における各アミノ酸(20種類)の相対頻度(上記の縦軸の値、スコアと呼ぶ)を20x20の行列式(PSSM)で表し、検査するアミノ酸配列を当てはめた時の各部位のスコアの平均をその配列の得点として、どこが相対的に切れやすそうかを予測するわけです。
しかし、この方法には2つの大きな欠点があります。一つは、既述のようにカルパインのアミノ酸の嗜好性はあまり強くないため、高い得点が出にくいことです。もう一つは、配列の文脈(コンテクスト、context)が考慮されていないことです。コンテクストとはどういう意味かというと、例えば、上図で、P2-P1/-P1’の3つのアミノ酸に注目してみます。P2はL, Tが、P1はT, Rが、P1’はS, A, Rがほぼ同じくらい高頻度に現れます。PSSM法によれば、これらの可能な12通り組み合わせによる配列LT/S, LT/A, LT/R, LR/S, LR/A, LR/R, TT/S, TT/A, TT/R, TR/S, TR/A, TR/Rは全て同じように切れやすいと予測されます。しかし、(実際には違いますが)もしもカルパインがLT/S, LT/R, TR/Aの3種だけをよく切って、他の9種の配列は全く切らない、という酵素であったとしても、PSSMはやはり上記の12種の配列を等しく良く切れるとしてしまいます。そのうちの9種は全く切れないにもかかわらずです。カルパインでは基質特異性の強いコンテクスト効果はまだ見つかっていませんが、他のプロテアーゼでは報告されているので、カルパインにも無いとは言い切れません。
これらの欠点を改善し、より正確な切断部位予測を可能としたのが、機械学習という手法です(下図)。特に、サポートベクターマシン(Support Vector Machine, SVM)と呼ばれる方法が、切断部位予測のような問題には最適と言われています。
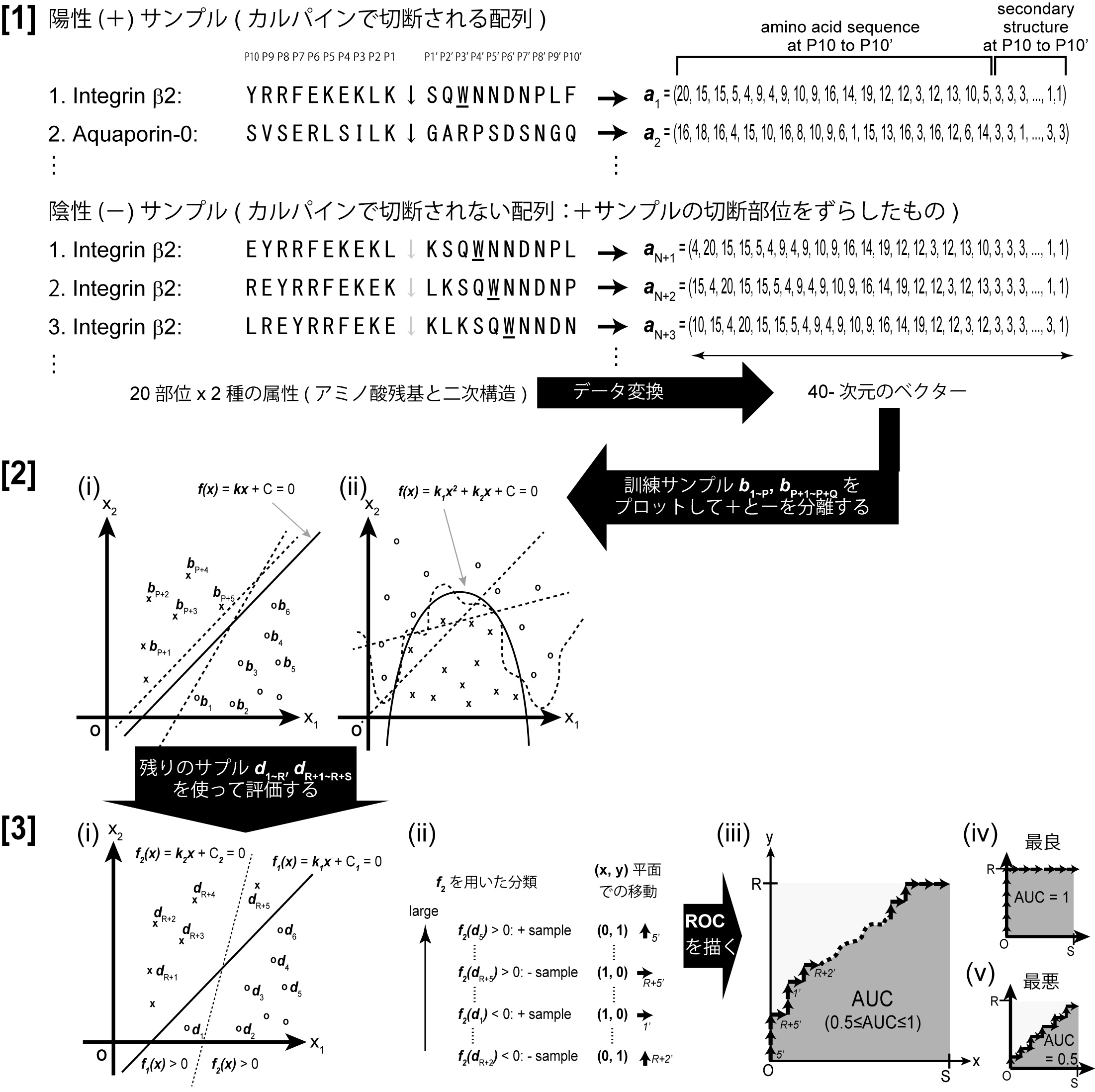
ここで、SVMで用いている数式は、数学的変換により核関数(kernel function、直感的には2つのベクターxとyがどれだけ近いかを数値化する関数でk (x, y)と表記されます。yが定値ならk (x)と書けます)を用いて簡単に表記・計算できます。SVMでは、用いる関数は一種類だけですが、データの種類(配列、二次構造、疎水性…)によってはそれぞれに違う関数を使って結合した方が有効な場合の多いことが知られています。そこで、複数の核関数を一次結合して、その重み付けをさらに学習させる方法を複合核関数学習(multiple kernel learning, MKL)と呼びます(下図)。ほとんどの場合単一SVMよりもMKLを用いた方が良い予測器を得られます。
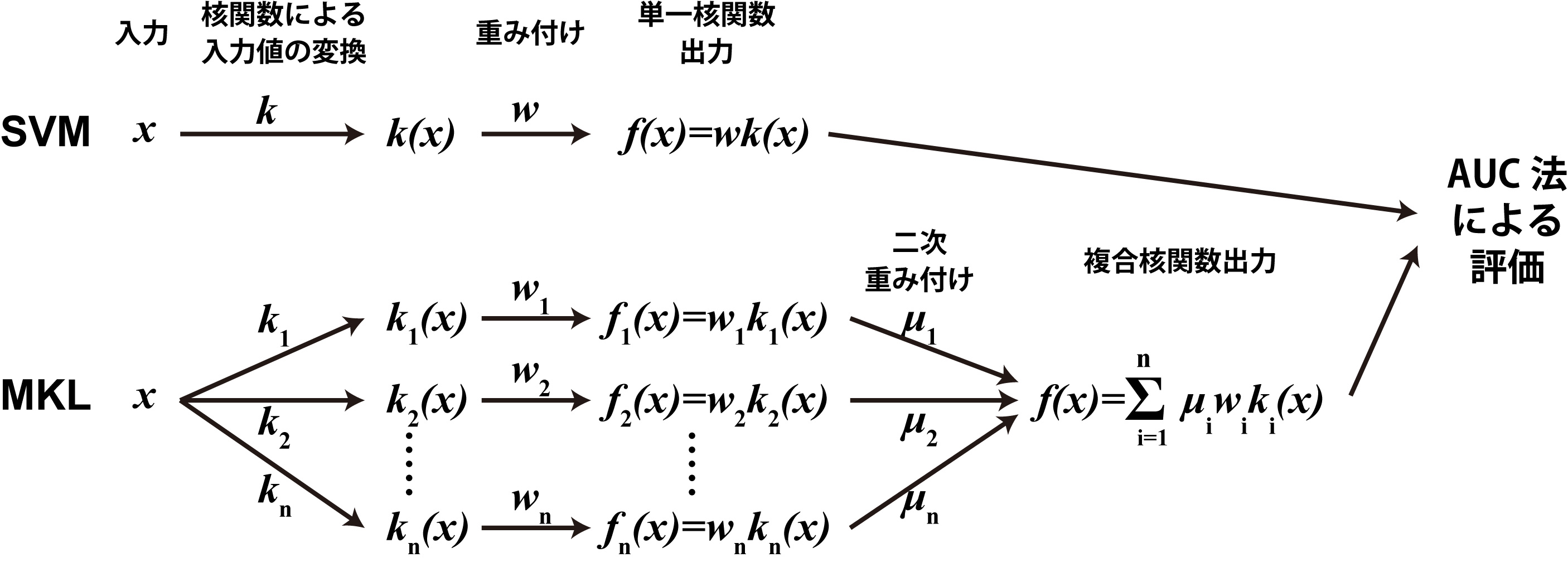
MKL予測器の利点の一つは、用いた複数の核関数に最適な重み付けが自動的にされるという点です。これは取りも直さず、切断部位の決定にアミノ酸配列のどのような特徴が重要かと言うことを示しています。カルパインの基質切断MKL予測器では、各部位のアミノ酸の種類、文字列、各部位の二次構造、各部位の溶媒接触性の4つの特徴に関してそれぞれに適当な核関数を用いた結果、重み付けは各々0.78, 0.59, 0.06, 0.18となり、意外にも二次構造よりも配列自体の方がずっと認識に重要であることが明らかとなりました。
また、上記220の切断部位は、CAPN1/S1(μ-calpain)とCAPN2/S1(m-calpain)とのデータが約半々のため、それぞれで予測器を作製した結果、前者と後者では切断部位周辺の認識する残基数に違いがあることが示唆されました。即ち、CAPN1/S1(μ-calpain)では、P14~P10’で、高いAUCが得られたのに対し、CAPN2/S1(m-calpain)では、P20~P20’のより広い範囲でたかいAUCが得られました。この結果は、前者よりも後者の方が、基質とより広い範囲で接触している可能性を示しています。さらに+サンプルを増やして検証したい興味深い示唆と言えます。
以上の詳細は、以下の原著論文か、 duVerle DA, Ono Y, Sorimachi H, Mamitsuka H (2011) Calpain cleavage prediction using multiple kernel learning. PLoS One 6:e19035.[http://dx.doi.org/10.1371/journal.pone.0019035] 以下の総説論文を参照下さい。 Sorimachi H, Mamitsuka H, Ono Y (2012) Understanding the substrate specificity of conventional calpains. Biol Chem 393:853-871.[ http://www.degruyter.com/view/j/bchm.2012.393.issue-9/hsz-2012-0143/hsz-2012-0143.xml](pdf(~7MB)はこちらから[Papers/BC_20120723_web.pdf])