Contents
- Research Summary of the Masai Laboratory
- Current Research Activities
- Future plans
- 最新研究成果(1) Rif1 preferentially binds to parallel-type and oligomerized G-quadruplex structures
- 最新研究成果(2) Regulation of chromatin structure by Rif1 through its binding to G4 at nuclear periphery
- 最新研究成果(3) Structures of DNA containing G4-forming sequences on duplex DNA
- 最新研究成果(4) Biochemical characterization of mammalian Rif1 protein
- 最新研究成果(5) DNA strand annealing activities of Mcm complexes
- 最新研究成果(6) Negative regulation of origin firing by Mrc1
- 最新研究成果(7) Regulation of initiation of DNA replication by Claspin
- 最新研究成果(8) Rif1 binds regulates replication timing through binding to G-quadruplex structures on the genome.
- Graphic representations of recent results
Research Summary of the Masai Laboratory
Precise duplication of genetic materials is central to the stable maintenance of genomes through generations. Defects in the genome copying processes would generate genomic instability which could ultimately result in various diseases including cancer. The goal of our studies is to understand the molecular basis of how the huge genomes are precisely replicated and the exact copies of the genetic materials are inherited to the next generation. Three billion base pairs of the human genome (2 meter long) are replicated with almost no errors during the 6-8 hrs time span of the cell cycle. This requires an extreme level of coordination of temporal and spatial arrangements of chromatin organization and signaling events for initiation of DNA replication (Masai et al. Ann. Rev. Biochem. [2010]). Our major projects include
1) Maintenance of genome integrity and its failure as a cause of diseases: Molecular dissection of cellular responses to replication stress, a major trigger for oncogenesis, and elucidation of mechanisms by which stalled forks are processed and the genome is protected from various insults, and of how the failure of this process leads to diseases and senescence (genetics and cell biology),
2) Chromosome dynamics that determines cell fate and regulates cell proliferation: Elucidation of mechanisms regulating temporal and spatial regulation of genome duplication as well as coordination of replication, repair, recombination and transcription. (genomics and molecular genetics; Yamazaki et al. Trends in Genetics [2013]),
3) Unraveling the universal mechanisms of origin firing and its regulation, and evolution of replication systems (molecular biology and enzymology), and
4) DNA replication and development: Understanding the roles of replication factors or replication timing regulation during development/ differentiation processes of various tissues and organs (mice engineering).
To achieve these goals, we are using E. coli, fission yeast, various mammalian cell lines, embryonic stem cells and model animals. We would like ultimately to apply the basic knowledge on the mechanisms of stable genome maintenance to the diagnosis and therapy of the relevant diseases including cancer. We are recruiting highly motivated and interested individuals who are communicative and can share excitement with us in the laboratory. We have had students from many foreign countries including Korea, Malaysia, Taiwan, China, Canada, Italy, France and Germany and have been excited to have many different cultures in our laboratory. Please feel free to contact us at any time through e-mail or by telephone. (https://www.igakuken.or.jp/genome/)
Current Research Activities
Our goal is to understand the molecular mechanisms underlying the faithful inheritance of genetic materials and stable maintenance of the genome. Toward this goal, we are studying various aspects of chromosome dynamics, with particular focus on regulation during S phase, using <i>E. coli</i>, fission yeast, and mammalian cells. We try to elucidate how chromosomes replicate and how the inheritance of the replicated chromosomes is regulated to enable stable maintenance of the genome through generations. Answers to these questions will shed light on how defects in these processes may contribute to the development of diseases, including cancers, and to senescence. It will also help to identify novel target proteins for cancer therapies.<br>Through these studies, we have come to realize the roles of non-B DNA, more specifically, G-quadruple structures. Recent numerous reports indicate the essential roles of various G4 structures in transcription, recombination, genome rearrangement, epigenome regulation and others.
Through analyzing the roles of G4 structures in replication initiation and chromatin regulation, we would like to disclose more general biological functions of G4 structures. <br>DNA replication needs to continue until the entire genome is replicated, once it is initiated. The block to ongoing DNA replication is a threat to the genomic integrity, and needs to be removed swiftly. Claspin/Mrc1 is a key protein that transmits the replication stress signal to the downstream effectors. We have discovered a novel function of Mrc1/Claspin in regulation of initiation through interaction with Cdc7 kinase. Furthermore, we have shown that Claspin is required also for different forms of stress. It is needed for cells to resume growth after serum starvation and for their responses to other stresses including osmotic shock and high temperature. We propose that Claspin may act as a general stress response mediator by linking stress-specific PIKK (PI3 kinase related kinase) and downstream effectors. We are interested in the in vivo roles of cell cycle/ checkpoint factors, and are making mutant mice in which these genes are conditionally knocked out in specific tissues/ organs by using Cre-loxP system. We are also developing novel anti-cancer strategies utilizeing cell cycle regulators as a target.
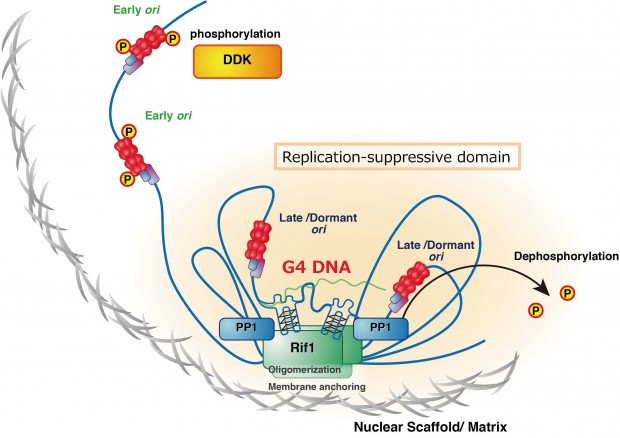
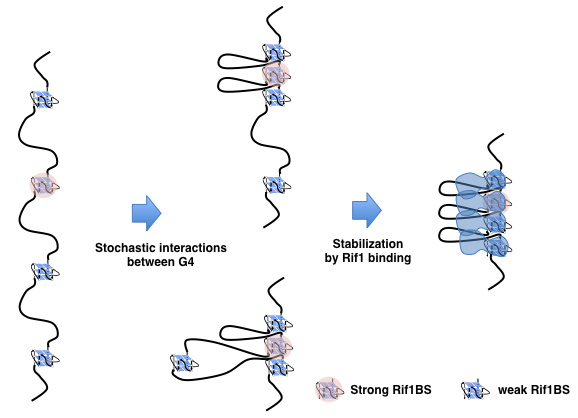
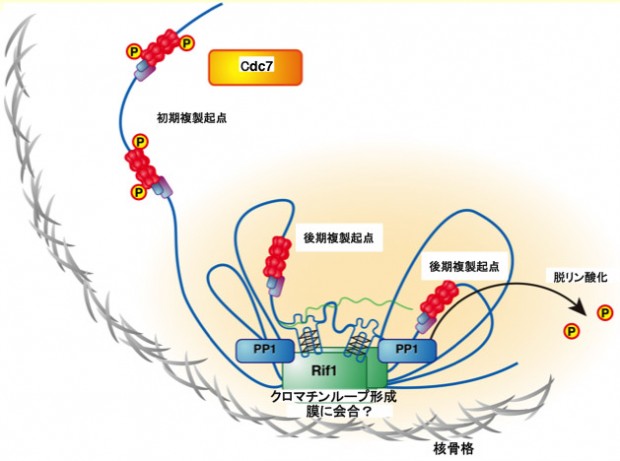
Fig. 1 Regulation of chromatin architecture for establishing replication timing domains. Rif1 facilitates chromatin loop formation through its binding to G4 structures present in the intergenic segments and its multimerization activitiy. This will generate chromatin architecture that may be related to the replication timing domain structures.
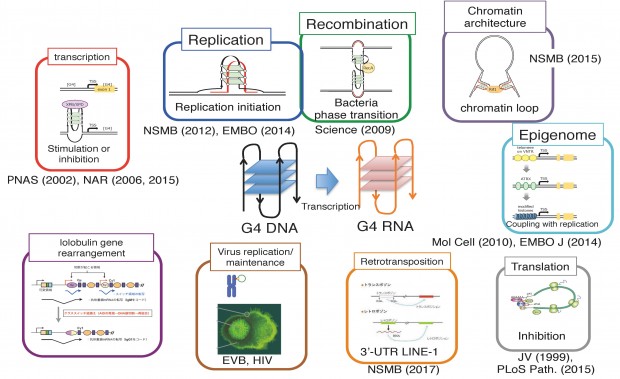
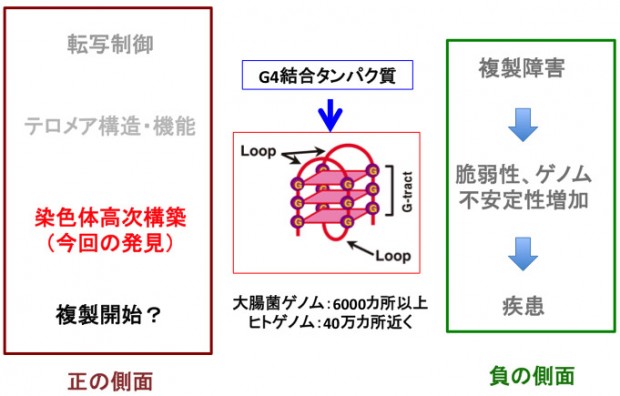
Fig. 2 Potential biological roles of G4. Increasing numbers of reports indicate the crucial roles of G4 in various chromosome transactions.
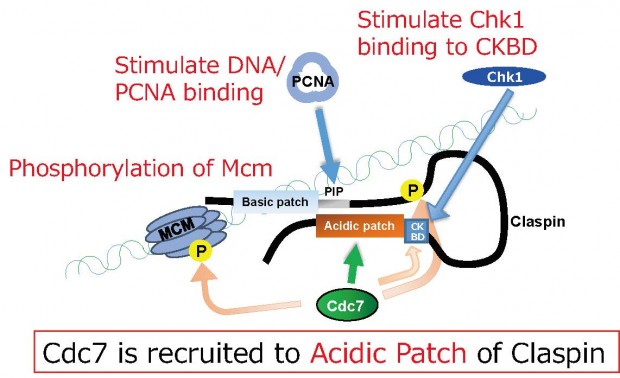
Fig. 3 Roles of Claspin and Cdc7 in regulation of replication initiation and replication stress responses. Cdc7 is recruited to the acidic patch segment of Claspin and facilitates Mcm phosphorylation. Cdc7 also facilitates DNA stress-induced Chk1 activation by promoting Chk1 binding to CKBD.
Future plans
In each cell cycle, three billion base pairs of the human genome are replicated within 6–8 hrs with amazing accuracy, while initiation takes place at tens of thousands of so called “replication origins”. Selection of replication origins for initiation is regulated by a number of factors, while a large fraction of origin selection may be determined stochastically. Yet, the initiation does take place at sequences that may be associated with some features. Our studies as well those from other groups suggest that G-quadruplex may be a candidate structure that may be associated with replication initiation. Our current goal is to reconstitute “human-like” replication system in E.coli with purified proteins on G4 sequences. <br> We have already indicated the importance of G4 in establishment of replication timing domain structures by showing that Rif1, a factor crucial for replication timing regulation, binds specifically to G4 DNA. We would like to understand molecular basis on how Rif1 facilitates chromatin loop formation by binding to G4. G4 is associated with other chromosome transactions as well, and a number of proteins bind to G4. We would like to clarify the modes and nature of interaction between G4 and its binding partners, elucidate the mode of actions of these proteins and ultimately obtain global views on potential biological functions of G4. <br>Replication factors are involved not only in the process of DNA replication but also in other aspects of chromosome functions. In order to clarify the organismal roles of replication factors, we target Cdc7, Claspin and other replication factors and generate genetically modified mice. Tissue/ organ-specific knock-out will reveal their novel functions, and will lead to understanding the pathology of human genetic disorders caused by mutations in these factors. <br>“Oncogenic stress” are known to induce replication stress, which inhibits the progression of replication forks. Thus, replication stress triggers the tumorigenesis process. The maintenance of replication fork integrity is central to the stable inheritance of genetic information through generations and to its preservation during the lifespan of an individual. Using mouse model systems as stated above, we will unravel the cellular and developmental roles of replication factors. Ultimately, we aim to apply our expertise on the molecular mechanisms of DNA replication and its physiology to the development of strategies for preventing and treating diseases caused by the loss of genetic integrity.
最新研究成果(1) Rif1 preferentially binds to parallel-type and oligomerized G-quadruplex structures
Article in Scientific Reports in press
論文題目:Rif1 promotes association of G-quadruplex (G4) by its specific G4 binding and oligomerization activities.
著者:Hisao Masai, Rino Fukatsu, Naoko Kakusho, Yutaka Kanoh, Kenji Moriyama, Yue Ma, Keisuke Iida, and Kazuo Nagasawa
研究成果の概要
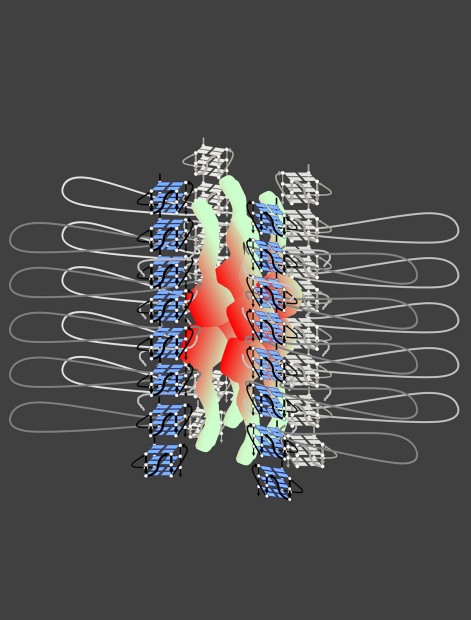
本論文では、進化的に保存された核因子Rif1と、その標的であるグアニン4重鎖(G4)構造について詳細な解析を行った。Rif1は、parallel あるいは hybrid/mixタイプのG4構造に、高い親和性で結合する。とくに、オリゴマー化したG4に選択的に結合する。Rif1は、8-16量体を形成し、複数のG4に同時に結合する。Rif1は、複数のゲノム上のG4構造と同時に相互作用することにより、クロマチンを束ね、染色体高次構造を形成する というモデルを提唱した。本論文では、Taz1とDNAとの相互作用についても、報告している。
図 Rif1はゲノム上に存在する複数のG4構造と同時に相互作用することによりクロマチンループを形成し、染色体高次構造形成の基盤となる。
最新研究成果(2) Regulation of chromatin structure by Rif1 through its binding to G4 at nuclear periphery
Article in Molecular and Cellular Biology 39(4). pii: e00364-18 (2019) Selected for cover figure
論文題目:Both a Unique Motif at the C Terminus and an N-Terminal HEAT Repeat Contribute to G-Quadruplex Binding and Origin Regulation by the Rif1 Protein.
著者:Kobayashi S, Fukatsu R, Kanoh Y, Kakusho N, Matsumoto S, Chaen S, and Masai H.
研究成果の概要
Rif1タンパク質は酵母からヒトまで進化的に保存され、染色体DNA複製開始の時間的タイミングと、核内での空間的配置を制御する。当プロジェクトでは以前、Rif1は代表的な非B型DNA構造であるグアニン4重鎖に結合することを報告した(Nature Structural and Molecular Biology 2015; J Biol Chem 2018)。今回、分裂酵母のRif1の詳細な変異体の生化学的、遺伝学的解析により、Rif1のC端ユニークモチーフおよびN端のHEATリピート構造の両者が独立にG4に結合すること、Rif1は12~16量体を形成すること、C端91アミノ酸のみで多量体形成できること、両モチーフが、Rif1による複製起点抑制活性に重要であることを示した。さらに、変異体のスクリーニングから、複製起点抑制能を喪失した新規変異体を単離した。これらの中にはクロマチン結合能を喪失しているものと、保持しているものが存在した。生化学的解析の結果にもとづき、Rif1による、クロマチンループ形成と、それによる複製開始を抑制するクロマチン構造の構築のモデルを提唱し、表紙に採用された(図参照)。この研究には、主に、当プロジェクトの修士2年の小林駿介氏、研究技術員の深津理乃氏、および研究員の加納豊博士により行われました。
図 Rif1タンパク質によるクロマチン高次構造形成のモデル
Rif1はC端G4結合ドメイン(赤)とN端HEATリピート構造(黄緑)から形成される。C端は8~16量体を形成するとともにG4構造に結合する。HEAT リピートも会合するとともに、G4構造と相互作用する。Rif1は、同時に複数のG4構造と相互作用できる。また電子顕微鏡での観察などに基づき、Rif1はN端とC端の両ドメインを介してゲノム上に散在するG4構造に結合し、これらを束ね、クロマチンループを形成する。Rif1は核膜と相互作用し、核膜近傍に局在しており、核膜の近傍でこのような構造を作ると考えられる。この構造は、TADやLADとも関連するとともに、複製開始に抑制的なクロマチンドメイン(中期~後期複製タイミングドメイン)を形成すると想像している。
最新研究成果(3) Structures of DNA containing G4-forming sequences on duplex DNA
Article in Journal of Biological Chemistry 293(44):17033-17049 (2018)
論文題目: Molecular architecture of G-quadruplex structures generated on duplex Rif1-binding sequences.
著者:Masai H, Kakusho N, Fukatsu R, Ma Y, Iida K, Kanoh Y, and Nagasawa K.
研究成果の概要
二本鎖DNA上のRif1結合部位が形成するグアニン4重鎖を含むDNA高次構造を詳細に解析した。その結果、G-rich鎖にG4構造が形成されるとともに、逆鎖にもS1感受性の構造が形成される。この構造は、G-rich鎖上のG4形成に依存しており、i-motifの形成を示す証拠は得られなかった。
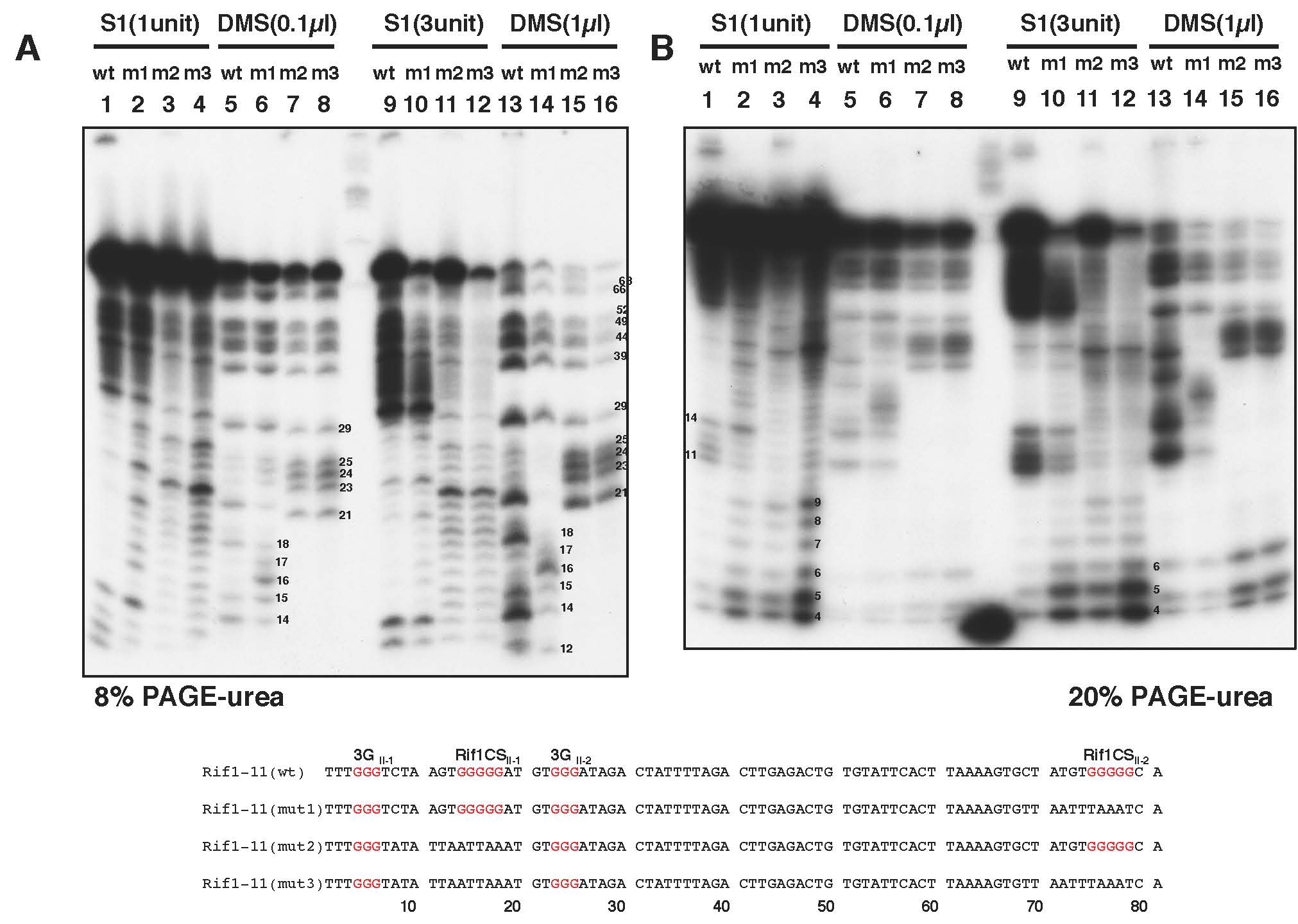 図 一本鎖Rif1BS DNA(野生型と、G4形成に影響をあたえる変異体)を熱処理した後に、S1ヌクレアーゼやDMSに対する感受性を解析した。G-tractsの変異により、別のG-traactを用いてことなる、G4構造が形成される可能性が示された。
図 一本鎖Rif1BS DNA(野生型と、G4形成に影響をあたえる変異体)を熱処理した後に、S1ヌクレアーゼやDMSに対する感受性を解析した。G-tractsの変異により、別のG-traactを用いてことなる、G4構造が形成される可能性が示された。
最新研究成果(4) Biochemical characterization of mammalian Rif1 protein
Article in Journal of Biological Chemistry 293(10):3607-3624 (2018)
論文題目: Oligomer formation and G-quadruplex binding by purified murine Rif1 protein, a key organizer of higher-order chromatin architecture.
著者:Moriyama K, Yoshizawa-Sugata N, and Masai H.
研究成果の概要
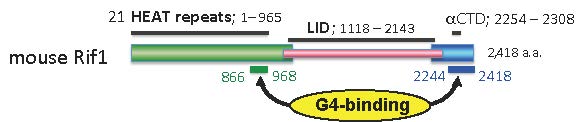
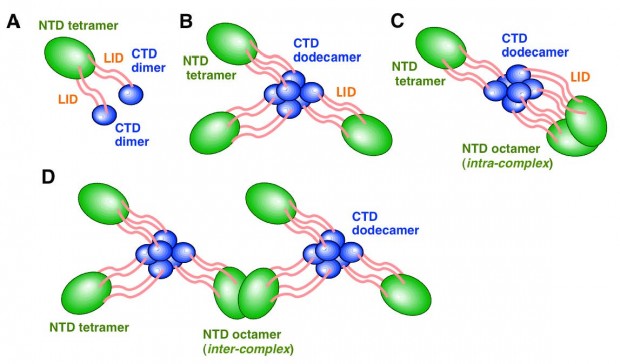
マウスRif1タンパク質を精製し、そのDNA結合能などについて詳細に解析した。C端およびN端HEAT リピートの両者にG4結合能力を有することを示した。また、Rif1は多量体を形成し、複数のG4構造に同時に結合することをしめした。
図1 マウスRif1タンパク質のG4結合ドメイン
図2 動物細胞Rif1タンパク質の多量体化のモデル
緑はN端HEATドメイン領域、青はC端のG4結合ドメイン、ピンクはIDP(天然タンパク質領域)を示す。
最新研究成果(5) DNA strand annealing activities of Mcm complexes
Article in Nucleic Acids Research 45(11):6494-6506 (2017)
論文題目:Potent DNA strand annealing activity associated with mouse Mcm2∼7 heterohexameric complex.
著者:You Z and Masai H.
研究成果の概要
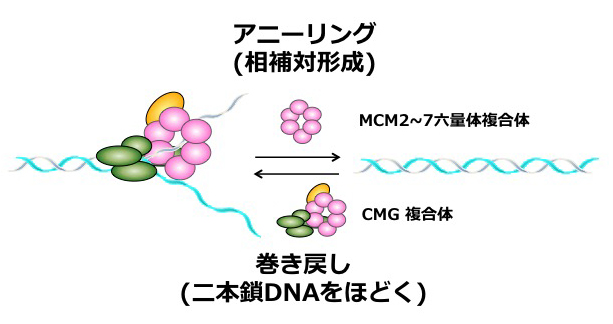
真核生物DNA複製で、二本鎖DNAがほどけてゆく反応は、もっとも重要な過程の一つです。この反応には、DNAヘリカーゼという酵素が重要な役割をはたします。Mcm2〜7六量体複合体は、DNA複製過程に必須な役割を果たす生物種に保存されたヘリカーゼの主要な因子であることが知られています。
私たちは、MCM複合体の一部であるMcm4/6/7複合体がDNAヘリカーゼ活性を持つことを世界に先駆けて発見し報告しました(You et al. 1999)。ところが、細胞内で重要な機能を果たすことがわかっているMcm2〜7六量体複合体は、これまで通常の条件下ではヘリカーゼの活性を示さないことから、未知の制御機構があると推測されていました。
その後、Mcm2~7六量体複合体が、他の複製因子である、Cdc45およびGINSと複合体を形成することにより、初めて活性のあるDNAヘリカーゼとなることが明らかとなりました。しかし、依然として、なぜMcm2〜7六量体複合体がヘリカーゼ活性を示さないかは不明でした。
今回、私たちは精製されたマウスMcm2〜7が、DNAヘリカーゼの逆反応である、二本の相補的な1本鎖DNAを会合させて、二本鎖DNAを形成するアニーリング(相補対形成)活性を持つことを発見しました。さらに、ヘリカーゼ活性によりはがされた 一本鎖DNAが、相補鎖形成することを阻害する条件では、Mcm2〜7は効率のよいヘリカーゼ活性を示すことを発見しました。このことからMcm2〜7は単独でヘリカーゼ活性を有するが、逆反応の活性のためにこれまで検出されていなかった可能性が示されました。
相補鎖形成反応は、DNA損傷などにより複製フォークが停止した時に生じる一本鎖DNAを二本鎖へと戻すことにより、複製フォークが壊れてしまわないようにする重要な役割があると考えられます。今回の発見は、Mcmが複製の過程で二本鎖DNAをほどいてゆくのみでなく、がんなどの疾患の原因となる、ゲノムの崩壊を防ぐという重要な働きをしていることを示します。
図 Mcm複合体は、強いアニーリング活性を有することが明らかとなった。このために、通常の条件下では、巻き戻し活性が検出されにくかった。一方、CMGヘリカーゼはアニーリング活性が抑制されているため、巻き戻し活性が検出されると考えられる。
最新研究成果(6) Negative regulation of origin firing by Mrc1
Article in Molecular and Cellular Biology 37(7). pii: e00355-16. (2017)
論文題目:Checkpoint-Independent Regulation of Origin Firing by Mrc1 through Interaction with Hsk1 Kinase.
著者:Matsumoto S, Kanoh Y, Shimmoto M, Hayano M, Ueda K, Fukatsu R, Kakusho N, and Masai H.
研究成果の概要
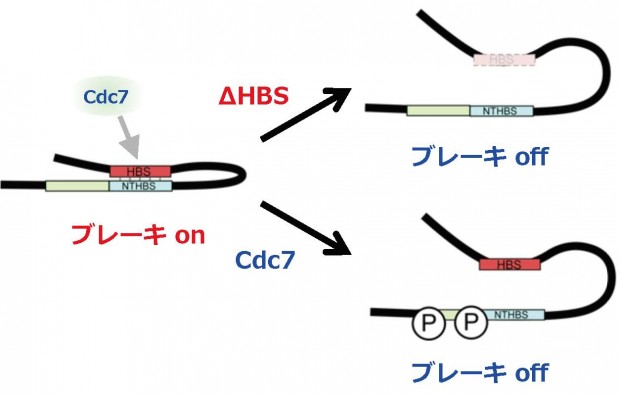
Mrc1は複製チェックポイントのメディエイターとして単離され、チェックポイント機能に依存して後期複製起点からの複製開始を制御(抑制)することが知られている。私たちは、Mrc1はチェックポイント非依存的に初期複製起点からの複製を制御することを見出し以前に報告した(Hayano et al. [2011]; Matsumoto et al. [2011])が、そのメカニズムは不明であった。今回私たちはMrc1は分子内相互作用を介してその分子形態を変化させ、複製開始のブレーキのon/offをすることを発見した。分子内相互作用に関与するHBSと名付けた領域には分裂酵母Cdc7キナーゼホモログであるCdc7が結合し、Mrc1のN端領域のリン酸化を介し分子内相互作用を解除しMrc1のブレーキを解除させる。このMrc1の分子内相互作用とそれによる分子形態変化を介したブレーキメカニズムは、チェックポイント非依存的な初期複製起点の選択と複製開始タイミングの制御に重要な役割を果たしていると考えられる。
図 分子内相互作用によるconformaton変換によるMrc1活性の制御
Mrc1は、通常、複製開始を抑制するブレーキonの形態を取っている(複製開始にhsk1機能を必要とする)。この時、Mrc1分子上のHBS(Hsk1 binding sequence)はN端近傍のNTHBSと分子内相互作用している。Cdc7キナーゼ(分裂酵母ではHsk1キナーゼと呼ばれる)はHBSを介してMrc1に結合し、NTHBSの近傍をリン酸化する。その結果、HBS-NTHBS間の相互作用が解除される。その結果、Mrc1のconformationが変化し、ブレーキoffの形態になる(右下;複製開始)。∆HBSはHBSの欠損のために、恒常的にブレーキoffのconformationをとるために、hsk1なしでも複製開始できるようになる(hsk1変異のバイパス)。
最新研究成果(7) Regulation of initiation of DNA replication by Claspin
Article in Nature Communications 7:12135. (2017)
論文題目:Claspin recruits Cdc7 kinase for initiation of DNA replication in human cells.
著者:Yang CC, Suzuki M, Yamakawa S, Uno S, Ishii A, Yamazaki S, Fukatsu R, Fujisawa R, Sakimura K, Tsurimoto T, and Masai H.
Review article in Current Genetics 63(5):813-818.
論文題目:Mrc1/Claspin: a new role for regulation of origin firing.
著者:Masai H, Yang CC, and Matsumoto S.
研究成果の概要
1 研究の背景
ゲノム複製は細胞の増殖にとって最も基本的な事象です。ヒト細胞は分裂するごとに、30億塩基対のDNAをほとんど誤りなくコピーし、全く同じゲノムDNAをもう1セット作り上げます。一方、複製の過程は6-8時間かかる長い過程ですが、その間に障害を受けて複製が止まったり、あるいはコピーの間違いを沢山起こすと、それはゲノムの変化として蓄積し、やがてがんやその他の疾患の原因となります。また、複製の開始・進行は、細胞周期と厳密に連動しており、その連動が破綻しても、ゲノムの変化が誘導されます。正常細胞においては、ゲノム変化が最小になるように、複製の過程は厳密に制御されています。一方、がん細胞では複製の制御に障害が生じることにより、ゲノムが”不安定”になる可能性が示唆されていますが、詳細は不明です。
2 研究成果の概要
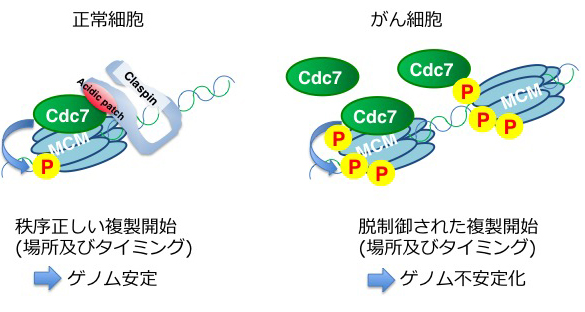
私たちは複製開始の引き金を引くCdc7キナーゼの研究を長年行ってきました。Cdc7はMcmというDNA二本鎖を開裂する酵素をリン酸化し複製開始を開始させることを発見し報告してきました。一方、Claspinは複製フォークの「見張り役」で、複製が止まったり、動きが悪くなったときにそれを見つけて修正する役割をはたします。今回、私たちはClaspinは複製フォークが形成される前にCdc7を呼び込む役割をすること、さらにClaspinはCdc7によりリン酸化され、その形態を変化させDNAとPCNAに結合できるようになり複製フォークにとりこまれることを発見しました。
さらに興味深いことに、がん細胞ではCdc7はClaspinがなくても複製開始に必要なリン酸化を行うことが明らかになりました。本研究の成果からClaspinは、Cdc7と連携して、細胞周期進行と連動した、秩序正しい複製開始と進行を保証することが明らかとなりました(図)。一方、がん細胞ではこの制御を失うことにより複製の開始と進行の脱制御が引き起こされ、細胞周期進行の異常、ゲノム不安定性亢進がもたらされまし。
3 発見の意義
Cdc7キナーゼは複製開始の引き金をひくが、今回の発見は、Claspinは複製フォークの進行のみならず、Cdc7キナーゼを呼び込む事により複製開始のタイミングと部位を制御することが明らかになりました。この発見は動物細胞のゲノム複製制御に新しい視点を加えました。一方、この制御システムはがん細胞では作動していないことも明らかになりました。本研究の発見は、がん細胞のゲノム不安定性の原因となる複製制御異常の一側面を初めて明らかにしたという点でも意義深いものです。
図 Claspinの複製開始における役割とがん細胞と正常細胞の違い
正常細胞:Claspinが結合するoriにおいてacidic patchにCdc7が呼び込まれ、Mcmのリン酸化をひきおこす。がん細胞;Claspinがなくても過剰に存在するCdc7によりMcmはリン酸化され複製が脱制御された状態で開始する。
最新研究成果(8) Rif1 binds regulates replication timing through binding to G-quadruplex structures on the genome.
Article in Nature Structural & Molecular Biology 22: 889-897 (2015)
論文題目:Rif1 binds to G-quadruplex and suppresses replication over a long distances.
著者:Kanoh Y, Matsumoto S, Fukatsu R, Kakusho N, Kono N, Renard-Guillet C, Masuda K, Iida K, Nagasawa K, Shirahige K, and Masai H.
研究成果の概要
ゲノムの中には通常のB型二重らせんDNA構造以外に多くのDNA特殊高次構造が存在する。グアニン4重鎖構造はその中でも特に多く存在する(大腸菌ゲノムに6000箇所、ヒトゲノムに35万箇所以上)。今回、ゲノム複製のタイミングを制御する因子,Rif1が、遺伝子間領域に存在するグアニン4重鎖構造に結合することによりゲノムの秩序正しい複製を可能にしていることを発見した。この因子は、グアニン4重鎖構造への結合を介して染色体のドメイン形成に関与し、複製や転写など染色体の多くの機能を制御する可能性がある。本発見はこれまで注目されていなかったゲノムのDNA特殊構造がゲノム維持、機能発現に大きな影響を与えることを示しており、今後、がんや、種々の疾患の原因解明にも新たなブレークスルーを与えることが期待される。
研究の背景:
私たちは、複製タイミングを決定する染色体ドメインを規定する因子の探索からRif1を同定した。Rif1は、酵母からヒトまで保存されており、その欠損はいずれの細胞においても複製タイミングの制御の喪失をもたらす。Rif1は、核膜近傍の核骨格構造において染色体に結合し、染色体機能ドメインを形成することにより、複製タイミングを制御する可能性を指摘した(図1)。さらにRif1欠損細胞の解析からRif1欠損は、転写プロファイルの変動(脱抑制)をもたらすことを見出した。また、Rif1はDNA切断修復過程にも関与する。このようにRif1は、染色体に結合し、クロマチンループの形成を介して、染色体の機能ドメインを制御する事により複製・転写・修復など種々の染色体の動態を制御すると考えられる。Rif1は核に存在し、染色体に結合することが知られているが、どのように染色体上に局在するかは知られていない。
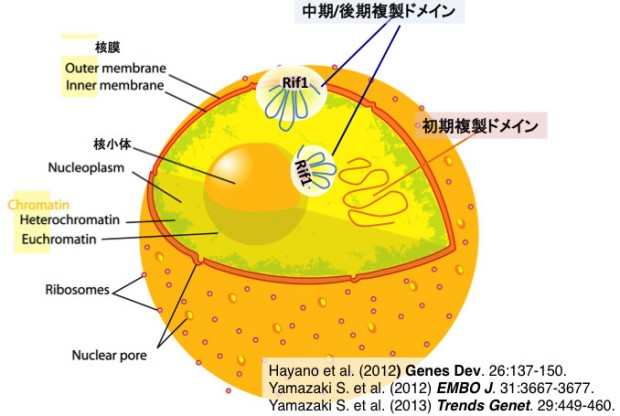
図1 Rif1は核膜上の核骨格結合領域に結合し、クロマチンループ構造の形成を介して中期・後期複製ドメインを形成する。
複製タイミングの研究についてはフロリダ州立大学のDave Gilbertらが先駆的な研究をしており、複製タイミングドメインは、TAD(染色体のトポロジーに関連したドメイン構造)に類似していることを示している(Nature 2015)。又複製タイミングの決定はG1期になされることをGilbertは報告したが、それを何が制御しているかは明らかでなかった。我々の発見からRif1がその主要な因子であることが示された。Rif1が、NHEJ(非相同末端連結)によるDSB(二重鎖DNA切断)修復に関与することは、国外の複数のグループが発見し報告した(Cell, Mol Cell, Scienceなど;2013)。またRif1の欠損により転写が影響を受けることも国外のグループにより報告された。Rif1のゲノムワイド結合部位とその解析については今日まで申請者のみが報告している。
G4構造については1980年代後半の発見以来、多くのその化学的、構造学的研究がされている。テロメア配列がG4構造を取りうることから、G4構造に結合する多くの化合物が同定、あるいは合成されている。東京農工大の長澤教授は、G4リガンドの合成、解析で世界をリードしているが、我々は彼らとの共同研究により、Rif1結合部位がG4構造を取りうる事を証明することができた。最近、これらのG4リガンドあるいはG4構造特異的な抗体を利用して、細胞内におけるG4構造の存在が示されている(Nature Chemistry, 2013他)。生物学的機能については、複製フォークの進行を阻害すること、またその阻害を一群のヘリカーゼがG4構造を解離することにより解除することが報告されている(Zakianら)。また遺伝子プロモーター上に存在し転写を阻害することも知られている。G4構造を取りうる配列は大腸菌ゲノム上に6000カ所、ヒトゲノムには40万カ所近くもあると推定されているが、そのゲノム機能における正の役割はこれまで不明であった。最近、ヒトゲノムの複製起点近傍にG4 構造をとりうる配列が濃縮されているという報告されたが、G4構造の複製開始における役割はまだ不明である。
結果の詳細:
本研究では、まず分裂酵母ゲノム上におけるRif1タンパク質の結合部位をChIP-Seq法*8により決定した(図2)。その結果、12.5Mbのゲノム上に35個の強い結合部位を同定しました。これらはいずれも遺伝子間領域に存在し、情報解析から、S期後期に開始する複製起点の近傍に存在することが明らかになりました。さらに結合部位の配列解析から、Gの連続配列を含む保存された配列を見出しました。この保存配列のほかにもRif1結合部位はGの連続配列を複数個含むことが明らかになりました。さらにゲノム上のRif1結合配列上の保存配列に変異を導入した結果、Rif1のクロマチン結合が局所的に消失するとともに、その近傍の複製タイミングが脱制御され、複製起点がS期初期に活性化されるようになった。これは、同定した保存配列がRif1のクロマチン結合とRif1の機能発現に重要な役割を果たしていることを示す。また、Rif1は結合部位の近傍100kbの領域のDNA複製の開始に抑制的な影響を与えることが明らかとなった。
図2 研究の流れ
次に、Rif1がどのようの保存配列に依存してRif1結合領域に結合するかを生化学的に解析した。精製したRif1タンパク質は、二本鎖DNAに結合するが、Rif1保存配列への依存性は観察されなかった。Gの連続配列が存在することから、G4構造を形成するかどうかを調べた。その結果、Rif1結合配列を熱変性して、再アニールすることにより保存配列に依存してG4様構造を形成することを確認した。さらに、精製したRif1タンパク質は、G4構造を形成した時に特異的に結合することを証明しました。
これらの発見は、細胞内でG4構造が実際に形成されており、それを特異的なタンパク質が結合し、重要な機能を果たしていることを遺伝学的に証明する(図3)。G4構造の染色体制御における普遍的な役割はこれまで全く不明だったが、今回の発見は、G4構造が染色体上の遺伝子間領域に存在し、それを特異的なタンパク質が認識、結合し、染色体の複製タイミングドメインを形成する可能性を示し(図4)、これまで未知であったG4構造の普遍的な生物学的意義の一端を明らかにするものである。
図3 Rif1は遺伝子間領域に存在するG4構造に結合し、クロマチンを束ねるとともに核膜近傍に複製抑制ドメインを形成する。
図4 グアニン4重鎖構造の生物学的意義:今回、染色体の高次構造の形成制御 という普遍的な機能の一端が明らかになった。
Graphic representations of recent results
生物の生存と遺伝において、遺伝情報の正確な倍加(DNA複 製)と娘細胞への伝達は最も基本的な生命現象の一つであ る。これが保証されないと、生命は存続できないことは言うま でもないが、その過程に異常が生じると、遺伝情報の誤りの蓄 積、ゲノムの不安定化がもたらされ、種々の疾患の原因となる。
私達は、このDNA複製の分子機構について主に3つの側面からアプローチしている。
(1)ゲノムの安定性維持機構とその破綻:増殖シグナル伝達、細胞周期は厳密に制御される。その破綻はただちに、細胞の異常増殖、種々の染色体異常等を引き起こし、がんなどの疾患、或は老化の原因となる。私達は、大腸菌・酵母・動物細胞(正常細胞、がん細胞、胚性幹細胞)・個体を用い、染色体DNA複製制御機構及び染色体の安定性維持機構を解析している。明らかになった分子機構に基づき、これらの過程が発生や分化の過程とどのように関連するか、又その異常がどのように種々の疾患を引き起こすか、更に、疾患の治療の新規分子標的の発見、制がん戦略・創薬の開発を目指す。
(2) 細胞の増殖、運命を決定する染色体ダイナミクスの制御機構(核内染色体高次構造の制御メカニズム):長大な染色体の複製は、厳密な時間的空間的プログラムに従って秩序正しく進行する。この過程は、核内染色体高次構造の制御と密接に関連し、転写・組換え・修復等の染色体ダイナミクスとも連動する。私達は、ゲノムワイドの染色体複製開始・進行の時空間制御機構のメカニズムを解明し、染色体高次構造を制御する新規のメカニズムを明らかにする。
(3)染色体複製の普遍的メカニズム・その起源と進化:大腸菌、分裂酵母、動物細胞更に変異マウスなど多様なモデル生物を用い、原核細胞からヒトまで保存された染色体複製の普遍的メカニズムの解明を目指す。特にゲノムのキャリアである染色体のB型らせん構造以外の特殊DNA構造・核内での染色体高次構築・配置がゲノム複製やゲノム機能発現をどのように制御するかに注目している。更に、得られた知見からDNA複製の起源と進化を考察する。
Cdc7キナーゼは複製開始に重要な役割をはたすリン酸化酵素である。分裂酵母を用いてCdc7キナーゼの複製への要求性をバイパスする因子を探索した結果、複製タイミングプログラムを制御するRif1を同定した。Rif1は分裂酵母のみならず動物細胞においても複製タイミングを制御することがその後の研究から明らかとなった。Rif1は、核膜近傍に局在し、クロマチンループを形成することにより、S期の中期・後期まで複製されない複製タイミングドメインを形成する可能性を指摘した。
![]¿Ïõï¿1-6-2014üè.pptx](https://www.igakuken.or.jp/genome/wp-content/uploads/2014/03/8d3a363f31f6a13e6fb8f8e83fd03073-620x464.jpg)
大腸菌など原核細胞では、通常染色体上のoriC部位からDnaAタンパク質に依存して開始する。この開始は大変効率がよく、塩基配列特異性も極めて高い。
![]¿Ïõï¿1-6-2014üè.pptx](https://www.igakuken.or.jp/genome/wp-content/uploads/2014/03/cSDR-620x464.jpg)
![]¿Ïõï¿1-6-2014üè.pptx](https://www.igakuken.or.jp/genome/wp-content/uploads/2014/03/50cfb7cf251ddd4664d2575e9c9a9386-620x464.jpg)
此れに対して、大腸菌ではDnaA-oriC非依存的な複製開始様式が存在することが知られている。この複製はRNA-DNAハイブリッド特異的にRNAを分解するRNaseH遺伝子の欠損株で観察され、転写とRecA組換え酵素およびPriAタンパク質に依存する。PriAタンパク質は複製フォークが停止した時にそれを認識し、複製の再会を促進することが知られている。DnaA-oriC非依存的な複製は、複製終結領域を中心に大腸菌染色体の多くの部位から開始する可能性が指摘されている。
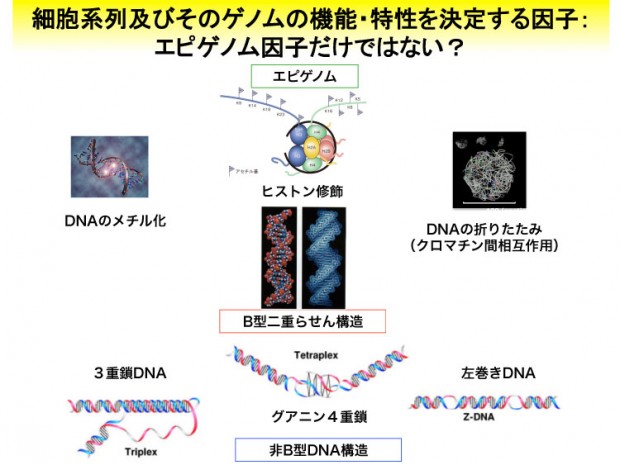
ゲノムを構成するDNAはおもにB型らせん構造からなるが、ゲノム上にはいわゆる非B型DNAが多く存在することが知られている。グアニン4重鎖はその中でも代表的なものである。ヒトゲノム上にも40万カ所近く存在する可能性があるグアニン4重鎖のゲノム維持における普遍的な意義はまだ不明な点が多いが、複製開始などへの関与が指摘されている。




![]¿Ïõï¿1-6-2014üè.pptx](https://www.igakuken.or.jp/genome/wp-content/uploads/2014/03/9e3045539b7c88aebdad52023fef129c-620x465.jpg)
![]¿Ïõï¿1-6-2014üè.pptx](https://www.igakuken.or.jp/genome/wp-content/uploads/2014/03/86edb6e3df1d2a2624433b6952971b89-620x464.jpg)
![]¿Ïõï¿1-6-2014üè.pptx](https://www.igakuken.or.jp/genome/wp-content/uploads/2014/03/ee7aab076d6cd2916b433540fb2fe85b-620x464.jpg)
