Contents
1 OVERVIEW
Our goal is to understand the molecular mechanisms underlying the faithful inheritance of genetic materials and stable maintenance of the genome. Toward this goal, we are studying various aspects of chromosome dynamics, with particular focus on regulation during S phase, using <i>E. coli</i>, fission yeast, and mammalian cells. We try to elucidate how chromosomes replicate and how the inheritance of the replicated chromosomes is regulated to enable stable maintenance of the genome through generations. Answers to these questions will shed light on how defects in these processes may contribute to the development of diseases, including cancers, and to senescence. It will also help to identify novel target proteins for cancer therapies. There are three major issues associated with genome DNA replication.
① Once and only once replication of a genome during a cell cycle and its strict coordination with mitosis.
② DNA replication during S phase is under sptiao-temporal regulation and the process is intimately related to epigenome maintenance and alterations.
③ DNA replication encounters various threats along the course of the process. Cells need to cope with these “replication stress” to ensure completion of the entire genome duplication without major errors.
The failures in ① and ③ are directly linked to increased genome instability and generation of cell populations of abnormal growth. In order to elucidate mechanisms associated with these issues, we are working on the following four major subjects.
1) How are the timing and nuclear localization of DNA replication determined, and how is it related to the processes of other chromosome transactions. We discovered crucial roles of Rif1, an evolutionally conserved nuclear factor, in genome-wide regulation of replication timing (Figure 1). We are particularly interested in how association of Rif1 with nuclear membrane contributes to regulation of not only replication timing but also other events such as DSB repair and transcription.
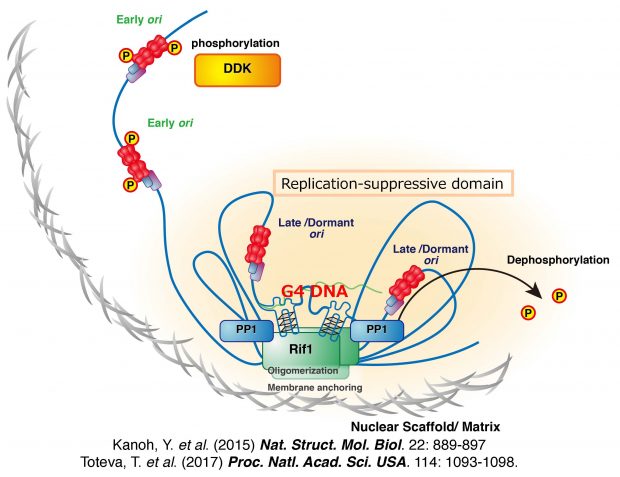
Figure 1 Regulation of chromatin architecture for establishing replication timing domains.
Rif1 facilitates chromatin loop formation through its binding to G4 structures present in the intergenic segments and its multimerization activitiy. This will generate chromatin architecture that may be related to the replication timing domain structures.
2) Biological functions of G-quadruplex, especially in regulation of DNA replication. Based on our findings, we have come to realize the crucial and ubiquitous roles of non-B DNA, more specifically, G-quadruple structures. Recent numerous reports indicate the essential roles of various G4 structures in transcription, recombination, genome rearrangement, epigenome regulation and others (Figure 2). Through analyzing the roles of G4 structures in replication initiation and chromatin regulation, we would like to disclose more general biological functions of G4 structures.
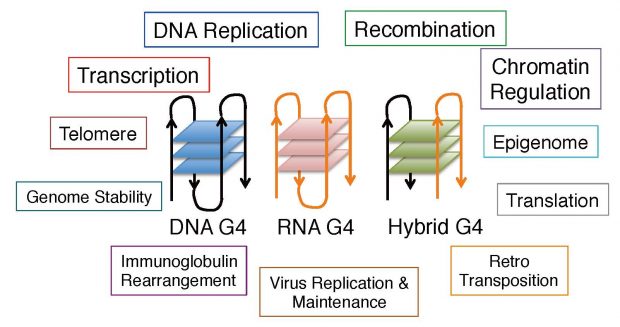
Figure 2 Potential biological roles of G4.
Increasing numbers of reports indicate the crucial roles of G4, formed on DNA, RNA and RNA-DNA hybrids, in various chromosome transactions.
3) Mechanisms of cellular responses to replication stress and how are they connected other diverse cellular stress responses. DNA replication needs to continue until the entire genome is replicated, once it is initiated. The block to ongoing DNA replication is a threat to the genomic integrity, and needs to be removed swiftly. Claspin/Mrc1 is a key protein that transmits the replication stress signal to the downstream effectors. We have discovered a novel function of Mrc1/Claspin in regulation of initiation through interaction with Cdc7 kinase. We also discovered a crucial role of Cdc7 in activating replication checkpoint through phosphorylation of Claspin (Figure 3). Furthermore, we have shown that Claspin is required also for cells to resume growth after serum starvation and for their responses to other forms of stresses including osmotic shock and high temperature (Figure 4).
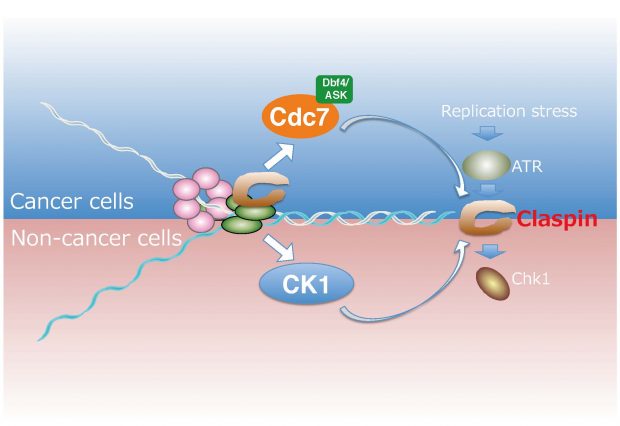
Figure 3 Differential mechanism of replication stress responses in cancer and non-cancer cells.
A conserved pathway induced by replication stress is drawn to the right corner of the figure. In this pathway, Claspin needs to be phosphorylated at a conserved CKBD motif to facilitate the binding of Chk1 kinase to Claspin. In cancer cells, Cdc7 is mainly responsible for this phosphorylation, whereas in non-cancer cells, CK1 (casein kinase 1g1) plays a predominant role. This differential mechanism can be exploited to develop a strategy for cancer cell-specific cell killing by targeting Cdc7 kinase.
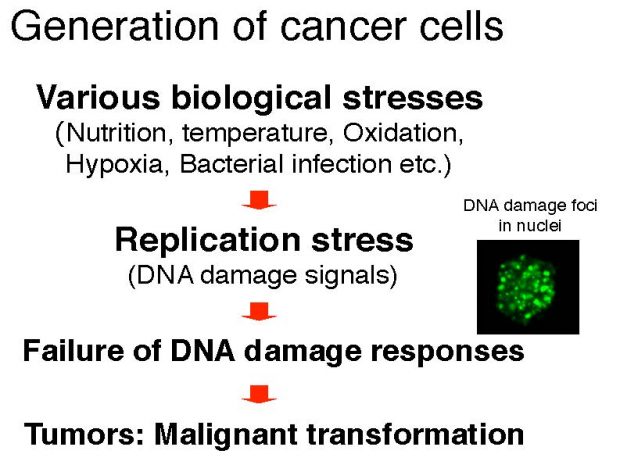
Figure 4 Formation of cancer cells.
Various cellular stresses could induce replication stress, thus activating Claspin-dependent checkpoint pathway. DNA damages are transiently induced by replication stress, but eventually removed by cellular DNA repair system. Once this cellular defense system is compromised, genome integrity is lost and cells are transformed into cancer state.
4) Roles of replication factors in development of individual organs and tissues and potential mechanistic diversification of replication systems. We are interested in the in vivo roles of cell cycle/ checkpoint factors, and are making mutant mice in which these genes are conditionally knocked out in specific tissues/ organs by using the Cre-loxP system (Figure 5). We are also developing novel anti-cancer strategies utilizing cell cycle regulators as a target.
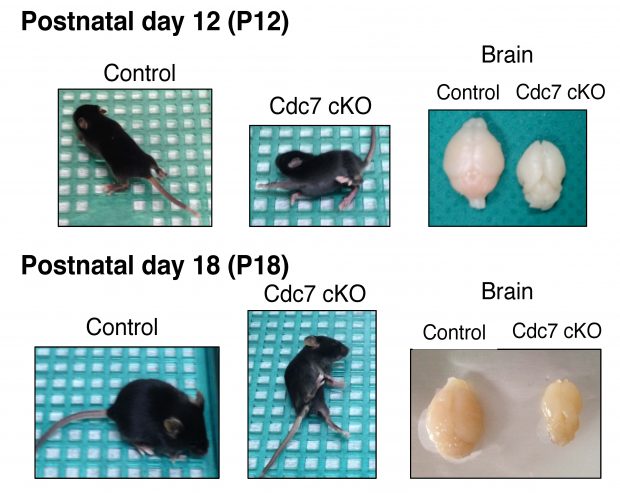
Figure 5 Cdc7 knockout in neural stem cells causes defect in brain formation and growth retardation after birth.
2 Short personal history of the leader
EDUCATION & DEGREE
1981 B.S., Dept. Biochemistry and Biophysics, Faculty of Science, University of Tokyo,
1986 Ph.D., University of Tokyo
EMPLOYMENT
1990 Assist. Prof., Dept. Mol. Dev. Biol., Inst. Med. Sci., Univ. Tokyo
1995 Assoc. Prof., Dept. Mol. Dev. Biol., Inst. Med. Sci., Univ. Tokyo
2000 Laboratory Head, Dept. Cell Biol., Tokyo Metropol. Inst. Med. Sci.
2004~ Visiting Professor, The University of Tokyo, Graduate School of Frontier Sciences
2011 Department Director, Dept. Genome Medicine, Tokyo Metropol. Inst. Med. Sci.
2012 Director of the Center for the Basic Technology Research Center, Tokyo Metropol. Inst. Med. Sci.
2015~ Vice Director General, Tokyo Metropol. Inst. Med. Sci.
2018~ Director General, Tokyo Metropol. Inst. Med. Sci.
EDITORIAL SERVICES
Editorial Board Member, Journal of Biological Chemistry (2009~2014)
Associate Editor, Genes to Cells (2009~)
Associate Editor, Journal of Biochemistry (2010~2012; 2017~)
Faculty1000 (Faculty Opinions) member (2011~)
Editor, Biochemical and Biophysical Research Communications (2014~)
AWARDS
2006: Tokyo Metropolitan Award
2012: A-IMBN (Asian International Molecular Biology Network) Arthur Kornberg Memorial Award
2017: 2017 Prize for Science and Technology (Research Category), the Commendation for Science and Technology by the Minister of Education, Culture, Sports, Science and Technology.
RECENT PUBLICATIONS (Excerpt)
- Masai, H. et al. Annual Rev. Biochem. 79, 89-130. (2010)
- Matsumoto, S. et al. J. Cell Biol. 195, 387-401. (2011)
- Hayano, M. et al. Genes and Development 26,137-150. (2012)
- Yamazaki, S. et al. EMBO J. 31, 3667-3677. (2012)
- Yamada, M. et al. Genes and Development 27:2459-2472. (2013)
- Kanoh et al. Nat. Struct. Mol. Biol. 22:889-897. (2015)
- Yang et al. Nat. Commun. 7:12135. (2016)
- Matsumoto et al. J. Cell. Biol. pii: e00355-16. (2017)
- Yang et al. E-life pii: e50796. (2019)
- Irie, T. et al. J. Med. Chem. 64(19):14153-14164. (2021)
- Yoshizawa-Sugata, Y. et al. J. Biol. Chem., 297(6):101367. (2021)
3 Current research subjects
(1) Universality and Diversity of DNA replication systems and their evolution
(2) Regulation of Spatio-temporal regulation of replication program through chromatin architecture and its spatial arrangement
(3) Dynamic formation and resolution of cellular G-quadruplex structures and their biological functions
(4) Cellular responses to replication stress and maintenance of genomic integrity: crosstalks with biological stress responses and relevance to cancer development
(5) Novel strategies for anti-cancer treatment targeting replication factors
(6) Roles of replication factors in development and functions of various organs and tissues
4 Current members of the lab (October, 2022)[Crick!]
Project Leader: Hisao Masai
| Current Laboratory Members | |||
| Sub leader | Hiroyuki Sasanuma | ||
| Staff Researchers | You Zhiying | Taku Tanaka | Yutaka Kanoh |
| Tomohiro Iguchi | Yoichi Tajima | ||
| Researchers | Sayuri Ito | Chi-Chun Yang | Kenji Moriyama |
| Students | Karin Hori (PhD 4th year) |
Tomoko Sagi (PhD 4th year) |
Shunsuke Kobayashi (PhD 2nd year; leave of absence) |
| Hao-Wen Hsiao (PhD 1st year) |
NGO THI TO TRINH (PhD 1st year) |
Yi Ding (PhD 1st year) |
|
| Manaho Sashida (Master 1st year) |
ZHEN WANXIN(Suzie) (Master 1st year) |
||
| Kosuke Yamazaki (BS 4th year) |
Kanna Hotta (BS 4th year) |
Ayaka Onuki (BS 4th year) |
|
| Itsuki Fukumoto (BS 3rd year) |
Keisuke Katsuda (BS 3rd year) |
||
| BAYRAK MELIH (Intership Student) |
WENXIN ZHENG (Intership Student) |
||
| Research Associates | Noaoko Kakusho | Rino Fukatsu | |
| Secretary | Akiko Minagawa | ||
| Laboratory assistances | Kazuto Takayasu | Yu Shiraki | |
| Visiting Scientists | So Maezawa | Makiko Hirano | Masayuki Yamada |
| Motoshi Hayano | |||
| Senior Visiting Scientists | Shoichiro Miyatake | Hiroyuki Kato | Takehiko Shibata |
| Foreign Visiting Scientist | Sebastian Jespersen Charlton (Denmark) |
||
5 Recent events (under construction in English; please see the Japanese HP)
2015年
2015年1月16日
津田ホールにて平成26年度 第7回 都医学研 都民講座「ゲノムの増えるしくみとその起源・進化:生命の起源から地球外可能性まで 」が開催。正井と東京薬大の山岸明彦教授が講演。
横浜情報文化センター 情文ホール にて、文部科学省新学術研究領域「ゲノムを支える非コードDNA 領域の機能」 主催 高校生向け公開講座「ゲノムの調べ」が開催される。2015年2月12日
銀座ミレジムにて旧友と再会。写真。2015年2月22日
東京マラソン開催。当研究室の新本美智枝さんが参加。3時間10分で完走。山崎君と応援にいきました。写真はこちら1。 2 3
(芝崎太先生も初マラソン 好記録おめでとうございます!)2015年2月25-26日
第一回医学研リトリート かずさアカデミアホールで開催されました。2015年3月26日講演
医療のパラダイムシフト~ 情報革命とバイオメディカル革命のシステム融合 ~ (かながわサイエンスパーク 3 階KSP ホール)2015年4月10日 Genes to Cells 編集委員会
2015年4月27日 及び 5月11日 東京理科大学 生物科学特別講義III(運河)
2015年5月14日 東京大学大学院 新領域創成科学研究科 メディカル情報生命専攻
発展講義Ⅳ(細胞高次機能の最先端)
「DNA複製制御の多様性、可塑性と進化そして疾患との関連」
2015年5月18~19日 BBRC Bangalore meeting (Bangalore, India)
May 18th (Monday) BBRC symposium at the National Centre for Biological Science (Title of the lecture: Biological roles of G-quadruplex structures )
May 19th (Tuesday) BBRC Board Meeting at The Taj West End
2015年5月13日、6月3,10日 東京大学 理学部 生物化学科「細胞分子生物学1」講義 (全3回)
2015年6月4~5日 大腸菌研究会(琵琶湖グランドホテル・京近江)
2015年6月21~26日 8th international fission yeast meeting in Kobe 情報はこちら。
2015年6月29~30日 韓国3大学ー医学研国際シンポジウム(東京都医学研)
2015年7月1~2日 CBSM2015 グラントエクシブ那須白河
2015年8月5~8日 新学術領域「非コードDNA」第9回領域会議(兼 国際会議)(淡路夢舞台)
2015年9月1~5日 Cold Spring Harbor Meeting “EUKARYOTIC DNA REPLICATION AND GENOME MAINTENANCE”。情報はこちら。
2015年9月11日(金)及び 9月18日(金) 3限~4限「生命科学特別講義」(学部との共通科目;南大沢)
2015年12月1~4日 BMB2015(第38回日本分子生物学会年会、第88回日本生化学会大会 合同大会)
神戸ポートアイランド
下記のセッションにおいて当研究室の発表があります。
セッション番号:『1W10』『ヘリカルリピートタンパク質の構造特性と細胞内機能』
日時:2015年12月1日(火) 9:00-11:30
会場:第10会場(神戸ポートピアホテル 南館 B1F トパーズ)
席数:約300席
発表予定者:森山 賢治
セッション番号:『1W2-p』『染色体の機能・構築原理』
日時:2015年12月1日(火) 14:00-16:30
会場:第2会場(神戸ポートピアホテル 本館 B1F 偕楽1)
席数:約340席
発表予定者:正井 久雄
セッション番号:『2W13』『非B型DNAの構造・生物学的意義とその生体制御への応用』
Structures and biological functions of non-B DNA and its application for manipulation of biological systems
ワークショップオーガナイザー 正井久雄 三好大輔
日時:2015年12月2日(水) 9:00-11:30
会場:第13会場(神戸ポートピアホテル 南館 B1F ルビー)
席数:約150席
発表予定者:加納 豊
セッション番号:『3W12』『DNA複製開始を制御する高次複合体ダイナミクス:多様性と普遍性』
ワークショップオーガナイザー 升方 久夫 片山勉
日時:2015年12月3日(木) 9:00-11:30
会場:第12会場(神戸ポートピアホテル 南館 B1F ダイヤモンド)
席数:約220席
発表予定者:山崎 聡志
2014年
2014年4月13-14日 新学術領域「ゲノム適応」班会議 (東京)
2014年4月19日 科学技術週間特別行事 夢祭エンス 医学研展示(日本科学未来館、江東区青梅)
2014年4月20日 第24回かすみがうらマラソン 兼 国際盲人マラソンかすみがうら大会 に参加(目標タイム4時間59分59秒–実際は生きて帰ること)。結果はこちら。
2014年5月16日 北京大学にて講演 (15-19日まで北京大学教授Daochun Kong博士を訪問)。こちらへ。
2014年5月21日 東京大学教養学部全学自由研究ゼミナール「生命科学の現在・パート16」講義「私の研究歴:太平洋を初めて渡って33年ーDNA複製の原理を探求して」こちらへ。
2014年6月5-6日 「第11回 21世紀大腸菌研究会」(岩手県盛岡市 つなぎ温泉「ホテル大観」)に参加(世話人)
2014年6月19-22日 延世大学・医学研 Joint Symposium (安東回口村(陶山書院があった村))
2014年7月14-16日 新学術領域「ゲノムを支える非コードDNA領域の機能」班会議(湯河原温泉ホテルあかね 〒259-0304 神奈川県足柄下郡湯河原町宮下705)正井発表は16日 11:25-11:50
2014年8月29-31日 九州大学 夏の学生研究発表合宿
2014年9月15-21日 Marco Foiani(Italy, Milan)教授の招聘により IFOM Foundation – F.I.R.C. Institute of Molecular Oncology Foundationで講演(演題:Regulation of DNA replication program in fission yeast and human cells)、及びSardinia島で開催される研究室ワークショップに参加。
2014年9月25-26日 大阪大学蛋白質研究所「染色体伝承の分子背景:複製から染色体分離まで」講演
2014年10月15-18日 第87回日本生化学会大会
片山 勉先生(九州大学)とともに、シンポジウム「ゲノムを支えるDNAヘリカーゼの動態を探求する新たな研究展開」をcoorgamnize (セッション番号:3S12a; 開催日:10月17日(金)9:00-11:30; 会場:第12会場[Room B-1])
KAISTのYeon-Soo Seo教授をこのシンポジウムには招聘しております。
2014年11月17-21日 9th 3R Symposium (Gotemba, Shizuoka)
Francesca Pisani教授(Istituto di Biochimica delle Proteine)及びFrancesca Carlomagno教授 (Universita’ degli Studi di Napoli Federico II)が、3Rに参加されます。3Rの前後に医学研でセミナーをしていただく予定です。
2014年11月25-27日 第37回日本分子生物学会年会 (パシフィコ横浜)
升方 久夫先生 (大阪大学)とともにワークショップ「ゲノムDNA複製制御のメカニズム:生物種を超えた統一像と多様性 (Conserved mechanisms and diversity of genome DNA replication: from bacteria to human)」をオーガナイズ。
セッション番号:『3W5』
日時:2014年11月27日(木) 13:15-15:45
会場:第5会場(パシフィコ横浜会議センター 3階 304)
2014年11月30-12月2日 2014 A-IMBN Governing Council MeetingおよびAnnual Conference (Henry Sy Auditorium, St. Luke’s Medical Center Global City, Manila, Philippines)に参加する。
2014年12月15日-12月17日
広島県宮島口にて第32回染色体ワークショップ・第13回核ダイナミクス研究会が合同開催。


