医学・生命科学全般に関する最新情報


新型コロナウイルスSARS-CoV-2のゲノムは30kbの1本鎖RNAから成っており、これは自然界で最も長大な1本鎖RNAゲノムです。
2本鎖の核酸は塩基対形成により制約を受け、その形態は限定されます。2本鎖DNAは、ほとんどの場合、B型右巻き二重らせん構造を形成します。一方、1本鎖RNAはその制約を受けないので、多様な高次構造を形成することが知られています。
SARS-CoV-2の遺伝子発現にはゲノム全長の転写(複製)とともに、discontinuous transcription(不連続転写)、また連続する異なる2つの読み枠(ORF1aとORF1b)から、2つの読み枠を連続して読むために起こる、frame shifting(読み枠移動)などの現象が知られています。これらの事象には、RNAの高次構造が重要な役割を果たすと想像されていましたが、多くは未解明でした。
最近Mol Cell誌に発表された論文(論文1)で、英国にケンブリッジ大学の研究者は、SARS-CoV-2の30kbの1本鎖RNAの高次構造を新しい方法で決定しました。
この論文の研究者らは以前に、COMRADES(Crosslinking Of Matched RNAs And Deep Sequencing)という方法を開発し、報告しました(論文2)。この方法はソラレンにより塩基対形成領域を標識し、proximity ligationによりRNA-RNA相互作用を網羅的に解析する方法です。
この方法をSARS-CoV-2 RNAゲノムに応用することにより、研究者は初めてSARS-CoV-2 RNAのRNA-RNA相互作用、さらにSARS-CoV-2 RNAと宿主RNAとの相互作用を検出することができました。
ORF1aからORF1bへの読み枠移動に~120ヌクレオチドのframeshifting element(FSE, 読み枠移動制御領域; ORF1aとORF1bの境界に存在)が、必要であることがわかっていました。今回の研究からFSE-archという、ORF1aの3’端と、ORF1bの5’端をつなげる1.5kbの高次構造が発見され、その中にFSEは埋め込まれて存在することが明らかとなりました(図参照)。
研究者は、今回の解析から、宿主のsmall nuclear RNAs(snRNAs)がウイルスのORF1aやORF1bと相互作用することも見出しました。また、Ribonuclease(RNase)mitochondrial RNA processing(MRP)RNAが不連続転写から生成されるsgmRNA(subgenomic mRNAs)の延長した3′端と相互作用することが明らかとなりました。RNase MRP RNAはrRNAの切断に関与することが知られています。
このように、ウイルスRNAゲノムの高次構造によりウイルスの遺伝子発現、複製が制御されており、RNA構造を標的とした新たな創薬の可能性が考えられます。
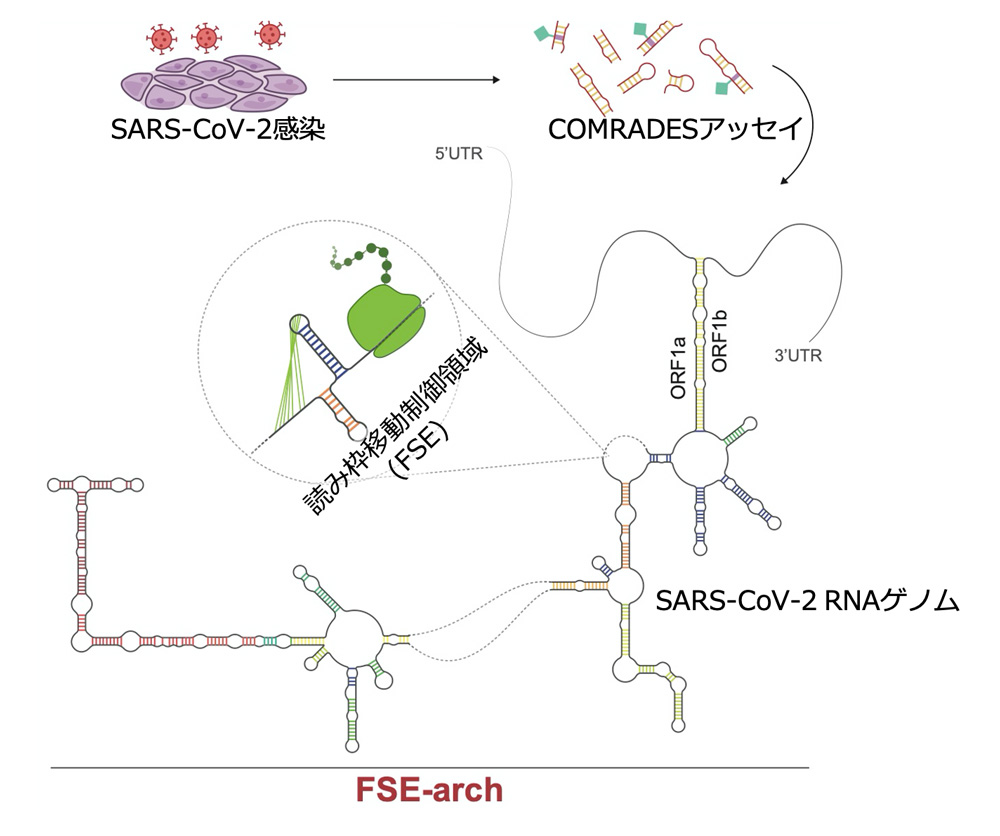
SARS-CoV-2感染細胞を用いて、細胞内のSARS-CoV-2 RNAゲノムのRNA-RNA相互作用を網羅的に解析した結果、遠く離れたRNA間、あるいは宿主のRNAとの相互作用の存在が明らかとなった。ORF1aとORF1bの境界に存在する読み枠移動制御領域(FSE)は、ORF1aとORF1bをコードするRNA領域が1500ヌクレオチドに渡って相互作用する町域(FSE-arch)の中に、埋め込まれていることが明らかとなった。(論文1から改変)